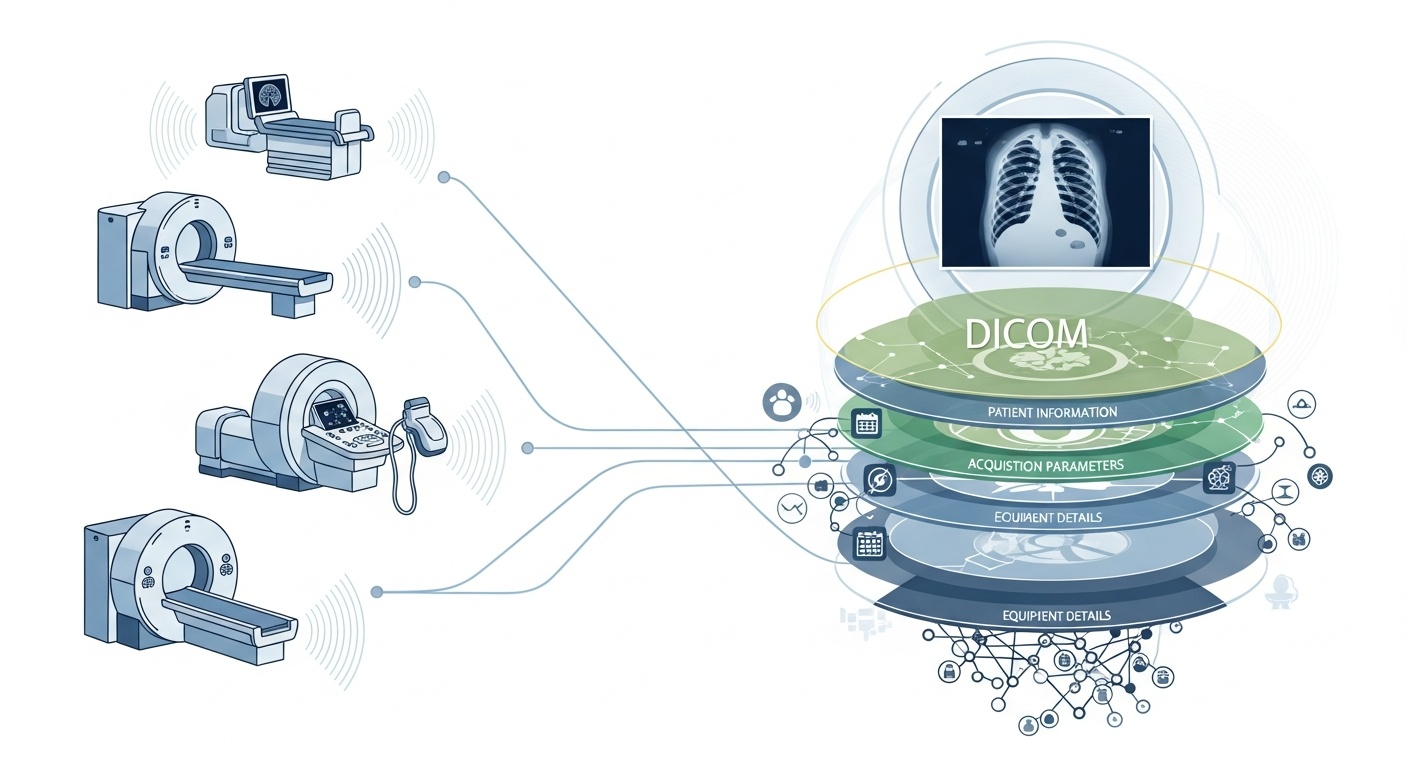
Sempre que uma equipa de radiologia examina um paciente, por exemplo, uma TC, RM ou ecografia, é gerada uma cascata de dados. O mais visível é a própria imagem, mas por trás dela existe uma rica camada de metadados: o quem, o quê, quando, como e onde do exame. Essa camada é regida pelo padrão DICOM (Comunicação e Imagem Digital em Medicina), um padrão de formato de imagem médica estabelecido pela National Electrical Manufacturers Association (NEMA) e pelo American College of Radiology (ACR) há décadas.
O que torna os metadados tão interessantes é o facto de serem estruturados, legíveis por máquina e notavelmente detalhados: definições do equipamento, parâmetros de aquisição, dados demográficos do paciente, IDs de estudo, e até códigos da instituição e detalhes do fabricante da modalidade. Essa riqueza é o que permite análises de big data, investigação a jusante, modelagem de IA e padronização de protocolos, se forem bem aproveitados.
No contexto de big data, não estamos apenas a falar de algumas dezenas de estudos de imagem. Estamos a falar de centenas de milhares, ou até milhões, de imagens entre modalidades, locais e fornecedores, com os metadados como a camada chave de indexação: o “quem & quando & como” de cada item. Sem utilizar eficazmente os metadados, corre o risco de ter um arquivo massivo de imagens, mas uma capacidade mínima de as consultar, comparar ou extrair conhecimentos das mesmas. Uma revisão recente afirma: “a maioria das informações armazenadas em arquivos PACS nunca mais é acedida”, uma oportunidade desperdiçada.
Digamos que está a realizar um estudo em vários locais de tomografias computadorizadas (TC) do pulmão para deteção precoce de enfisema. Irá querer selecionar exames com base em parâmetros: fornecedor/marca/modelo do scanner, espessura do corte, kernel de reconstrução, idade do paciente, intervalos de datas, talvez até parâmetros de dose. A maioria destes são campos de metadados, não dados de píxeis. Extrair essas tags permite-lhe construir o coorte, excluir exames incompatíveis (por exemplo, cortes demasiado espessos) e garantir a comparabilidade.
Os metadados permitem monitorizar o próprio processo de imagem: a instituição está a usar o protocolo correto? Os parâmetros de aquisição estão a desviar-se ao longo do tempo (por exemplo, campo de visão, tempo de injeção de contraste)? As definições do fornecedor são consistentes? Num mundo de big data onde ocorrem milhares de exames por dia, não pode confiar na verificação visual humana. Precisa de análises sobre metadados. Muitos sistemas PACS subexploram os metadados por esta razão.
Se construir um pipeline de IA ou radiómica, não pode tratar cada imagem como intercambiável. Os metadados tornam-se uma variável de controlo integral: as características de entrada incluem frequentemente modalidade, kVp (pico de quilovoltagem), fabricante, kernel, espessura do corte, e até a data ou o hospital podem importar (desvio de domínio). Estes campos de metadados ajudam a gerir preconceitos, harmonizar dados e anotar imagens com contexto. Muitos investigadores chamam aos metadados “tão importantes quanto os dados de píxeis.”
Big data significa escala. Isso implica fontes variadas, múltiplos fornecedores, diferentes instituições e formatos heterogéneos. Os metadados DICOM são a “linguagem” padronizada que ajuda a unificar a camada de metadados, permitir pesquisa/indexação, garantir interoperabilidade e construir arquiteturas escaláveis (Cloud PACS, arquivos federados). Mas a implementação importa. O mesmo estudo PMC descobriu que muitos sistemas não exploram totalmente o padrão.
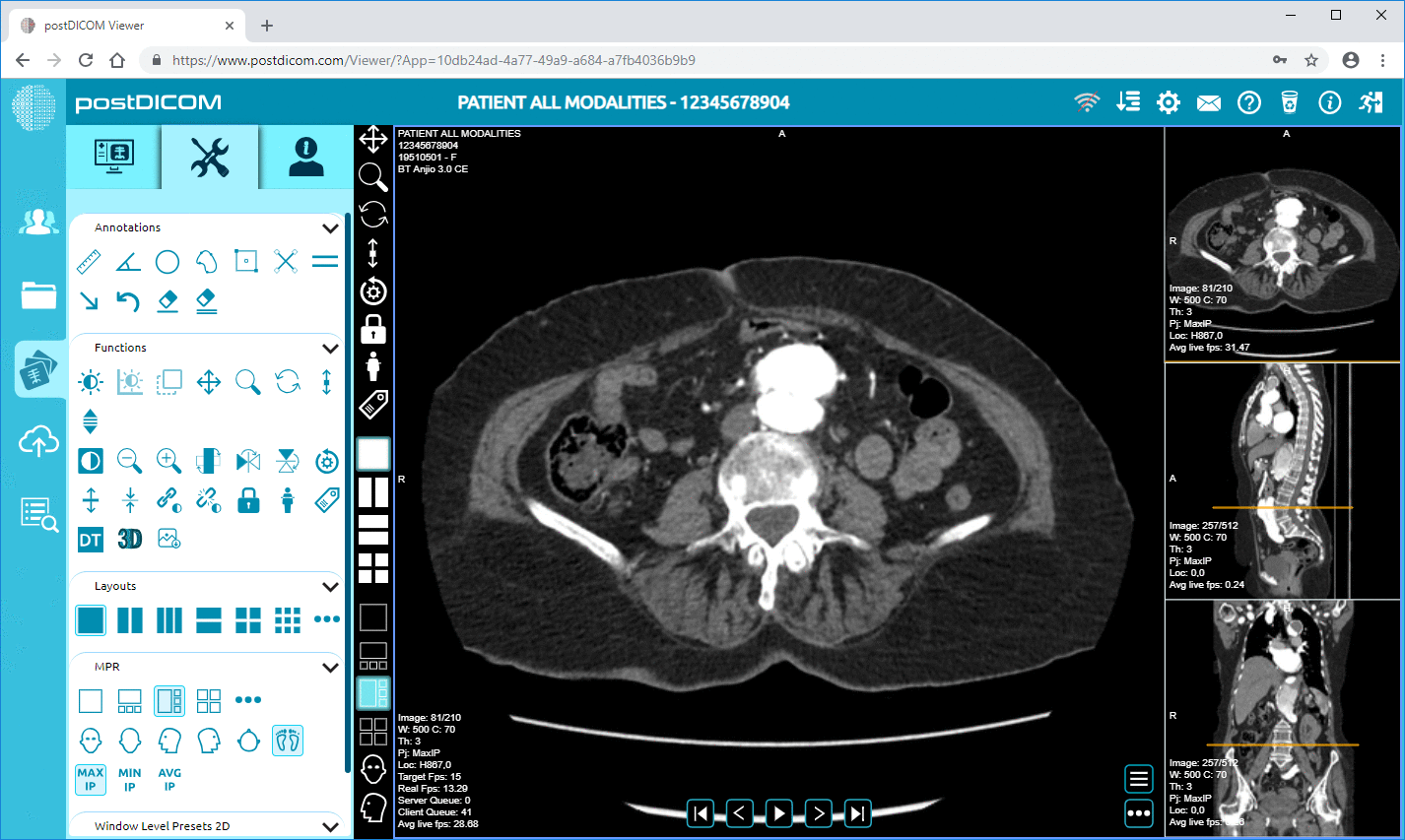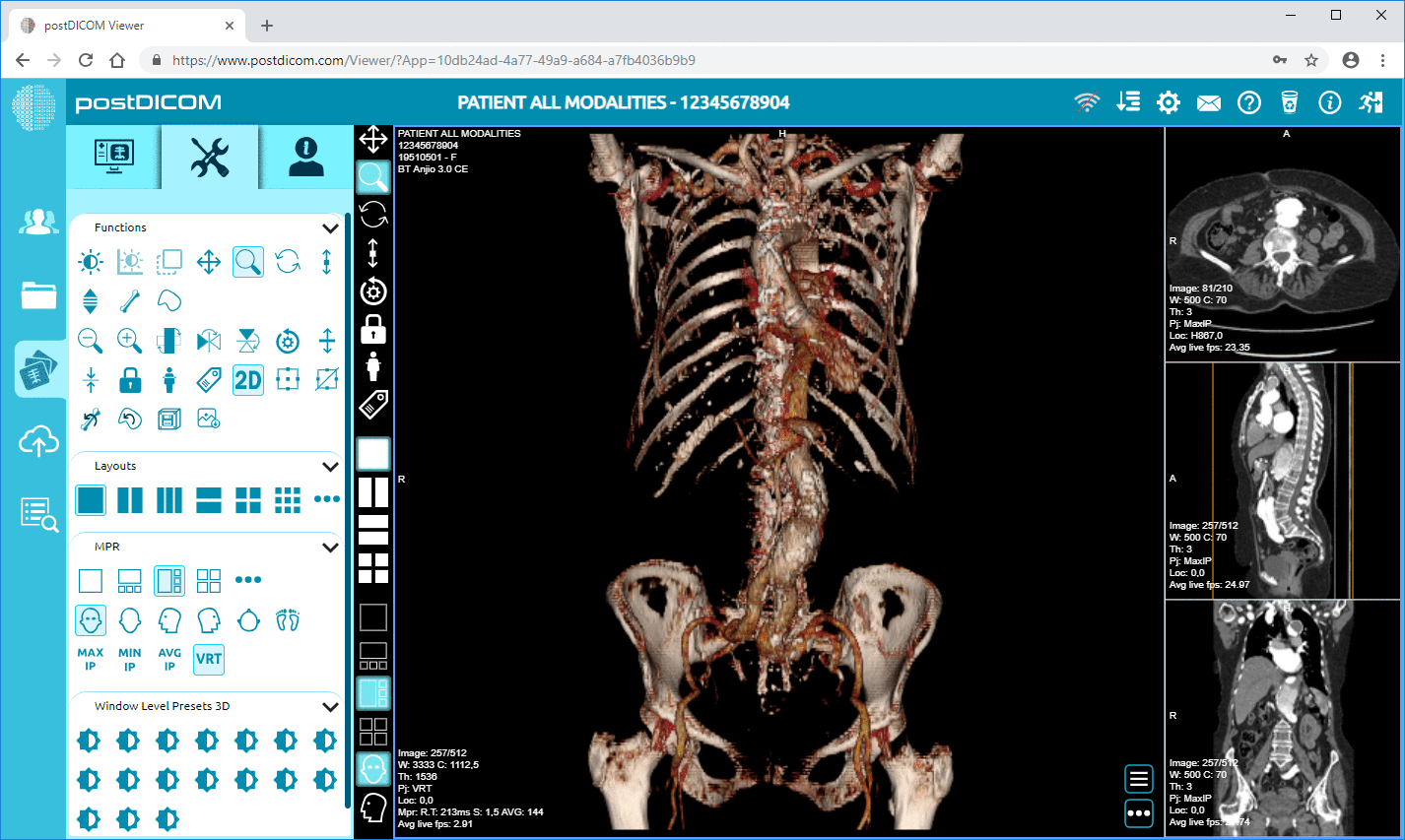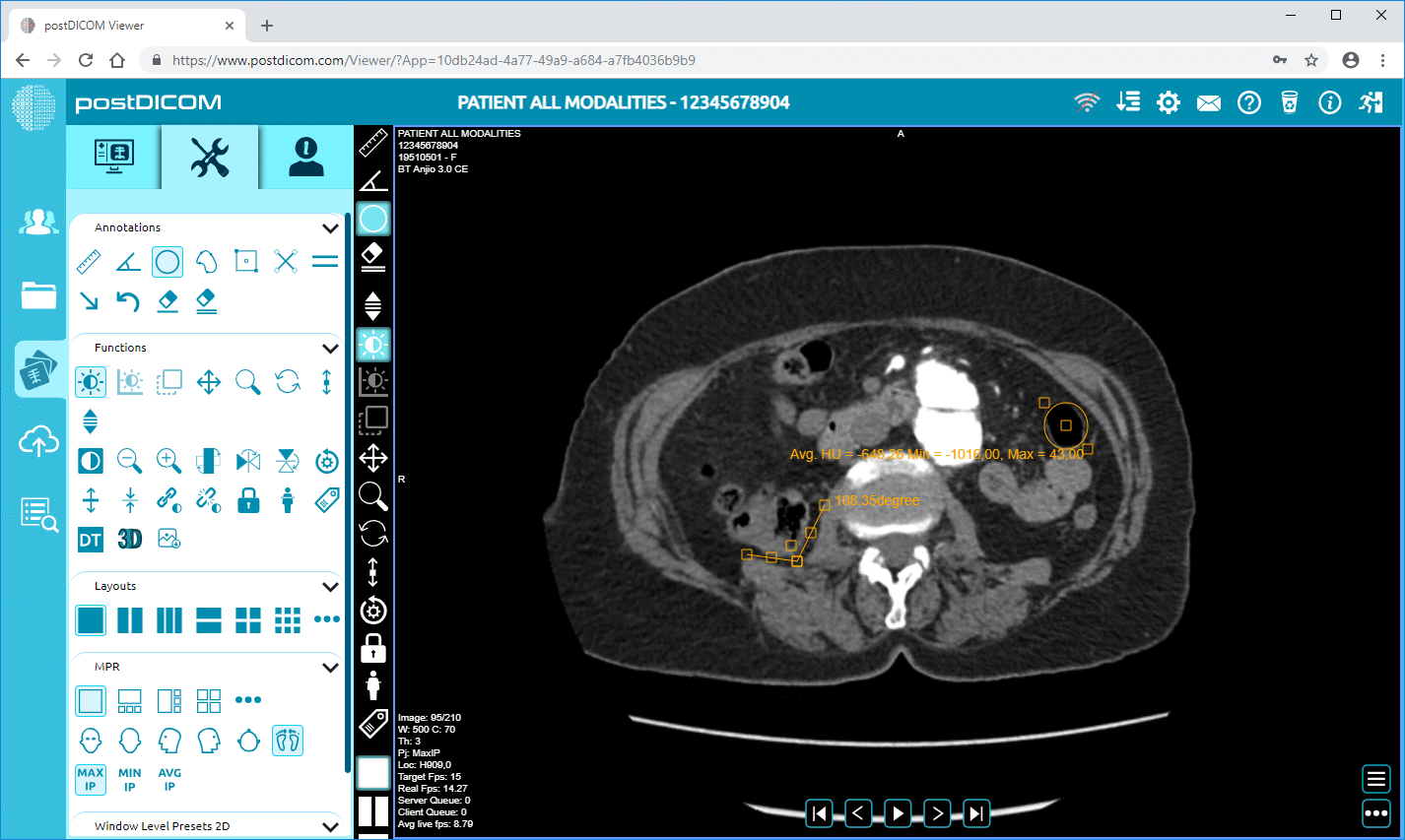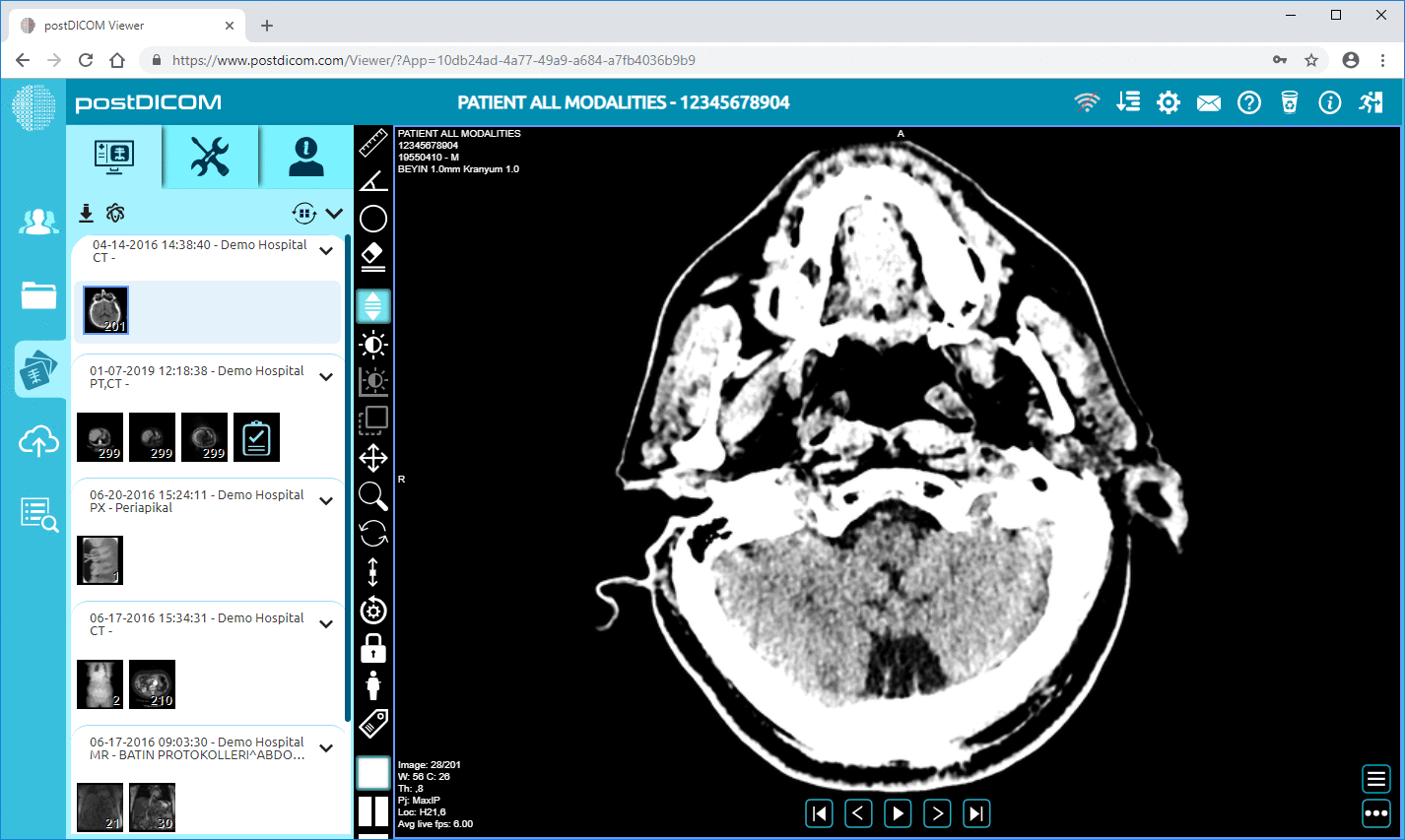
O PostDICOM oferece um PACS (Sistema de Arquivo e Comunicação de Imagens) baseado na nuvem para armazenar, visualizar e partilhar estudos de imagem e documentos clínicos. Algumas características chave relevantes para metadados do PostDICOM:
• Suporte Para Tags e Descrições DICOM: A nossa biblioteca de recursos lista “Modalidade e Tags DICOM”, permitindo aos utilizadores aceder a listas de tags e descrições.Modalidade e Tags DICOM
• Integração API / FHIR: Suporta interfaces API e FHIR (Recursos de Interoperabilidade Rápida em Saúde), permitindo que os metadados sejam consultados programaticamente, integrados com outros sistemas e analisados.
• Escalabilidade na Nuvem e Partilha Multi-local: A partilha entre pacientes, médicos e instituições; a escalabilidade ilimitada significa que os pipelines de big data tornam-se viáveis.
• Processamento Avançado de Imagem e Suporte Multi-modalidade: Embora isto diga respeito a píxeis, o suporte de modalidades como PET–CT e multi-série significa que os metadados são substanciais (valores SUV, volumes de fusão, tipo de modalidade) e estão disponíveis para análise.
Utilizar uma plataforma como o PostDICOM permite-lhe aproveitar os metadados através de fluxos de trabalho estruturados, APIs e arquitetura na nuvem.
Eis como estruturar o fluxo de trabalho desde arquivos brutos até conhecimentos prontos para análise.
 - Created by PostDICOM.jpg)
O primeiro passo para aproveitar os metadados DICOM para análise é a extração e normalização. Bibliotecas como o pacote Python de código aberto PyDicom são comummente usadas para analisar ficheiros DICOM e extrair tags relevantes, incluindo linhas e colunas de imagem, kernels de convolução e parâmetros de aquisição específicos da modalidade.
Lidar com a heterogeneidade é crucial, uma vez que diferentes fornecedores usam frequentemente tags privadas ou implementações não padrão. Análise robusta, lógica de fallback e tabelas de mapeamento de tags abrangentes são necessárias para garantir a consistência entre conjuntos de dados.
Uma vez extraídos, os metadados devem ser normalizados e mapeados para ontologias e estruturas padrão, tais como códigos de modalidade, nomes de fornecedores, categorias de espessura de corte e formatos padronizados de data e hora.
Finalmente, os metadados estruturados devem ser armazenados num ambiente de big data, como uma base de dados relacional, armazenamento NoSQL ou data lake colunar, com indexação para permitir consultas rápidas e eficientes.
Uma vez extraídos, os metadados devem passar por garantia de qualidade para assegurar precisão e fiabilidade. Campos em falta ou inconsistentes, como valores de espessura de corte em branco, rótulos de modalidade inconsistentes ou UIDs de Instância de Estudo duplicados, precisam de ser identificados e corrigidos.
Privacidade e anonimização são também críticas nesta fase, uma vez que os metadados contêm frequentemente informações pessoalmente identificáveis, incluindo nomes de pacientes, IDs e datas; ferramentas e protocolos de desidentificação são essenciais.
Manter trilhas de auditoria abrangentes é outra prática importante, documentando quando os metadados foram extraídos, quais as versões do analisador utilizadas e quaisquer erros encontrados durante o processo.
As políticas de governança devem também definir campos obrigatórios e fornecer orientação sobre como lidar com conjuntos de dados legados ou incompletos para garantir que as análises a jusante sejam precisas e conformes.
O próximo passo é a indexação orientada por metadados e a engenharia de características, que transforma metadados brutos em informação acionável.
Isto envolve a criação de índices e filtros que permitem aos investigadores e analistas consultar conjuntos de dados específicos, por exemplo, recuperar todas as tomografias de tórax com espessura de corte inferior a 1,5 milímetros de um determinado fornecedor num intervalo de datas específico.
A engenharia de características baseia-se nisto ao combinar campos de metadados como fornecedor, modelo, data de aquisição, espessura do corte, kernel de convolução, protocolo de contraste, região do corpo, dose de radiação e ID da instituição em variáveis estruturadas adequadas para análise.
Os metadados também podem ser ligados a conjuntos de dados clínicos, conectando dados de imagem a resultados de pacientes, diagnósticos ou tratamentos. Esta ligação permite uma visão mais holística dos dados de imagem e do seu contexto clínico.
Uma vez indexados os metadados e as características engenhadas, a análise e a geração de conhecimento tornam-se possíveis.
A análise descritiva pode revelar volumes de estudo por modalidade, fornecedor ou região, rastrear tendências nos parâmetros de aquisição e destacar erros ou inconsistências nas práticas de imagem. A análise comparativa permite a avaliação de protocolos de aquisição entre instituições, deteção de desvios e identificação de exames atípicos que podem requerer atenção especial.
Para aprendizagem automática e aplicações de IA, os metadados são essenciais para controlar o desvio de domínio, garantindo que os conjuntos de dados de treino e teste são estratificados adequadamente, e combinando características baseadas em píxeis com variáveis de metadados estruturadas. Os painéis operacionais podem então aproveitar estes dados para monitorizar a carga de trabalho, avaliar métricas de garantia de qualidade e assegurar a conformidade do protocolo entre locais.
Finalmente, o feedback e a melhoria contínua completam o ciclo de vida dos metadados. Conhecimentos derivados da análise podem informar o refinamento dos protocolos de aquisição e a padronização dos fluxos de trabalho para melhorar a qualidade geral dos dados.
Novos estudos de imagem e metadados devem ser continuamente ingeridos, com monitorização do desempenho do armazenamento de metadados, tempos de consulta e integridade dos dados. As lições aprendidas devem ser arquivadas para capturar campos de metadados preditivos, abordar lacunas ou erros recorrentes e melhorar as práticas de governança.
Esta abordagem iterativa assegura que os pipelines de metadados permaneçam robustos, escaláveis e valiosos para pesquisas futuras, aplicações de IA e tomadas de decisão operacionais.
• Variabilidade de Fornecedor/Instituição: Tags privadas ou interpretações vagas dos padrões.
• Metadados em Falta ou Corrompidos: Estudos mais antigos podem ter cabeçalhos incompletos.
• Privacidade de Dados e Anonimização: PHI deve ser desidentificado para investigação multi-local.
• Escala e Desempenho: Milhões de imagens requerem processamento e armazenamento eficientes.
• Desvio/Viés de Domínio: Fornecedores/protocolos dominantes podem distorcer os modelos de IA.
• Questões Regulatórias e de Conformidade: Implementações multi-regionais podem envolver HIPAA, RGPD ou regulamentações locais.
Os metadados DICOM são o esqueleto oculto da análise de imagem. Plataformas como o PostDICOM ilustram como transformar um arquivo fragmentado de ficheiros DICOM num ecossistema pesquisável, escalável e orientado por metadados. Se quiser explorar o PostDICOM, encorajamo-lo a obter o nosso teste gratuito de 7 dias.
|
Cloud PACS e Visualizador DICOM OnlineCarregue imagens DICOM e documentos clínicos para os servidores PostDICOM. Armazene, visualize, colabore e partilhe os seus ficheiros de imagiologia médica. |